9. La Medicina Personalizzata oltre i bersagli molecolari: dalla parte dei Pazienti
a cura di P. Marchetti – Fondazione per la Medicina Personalizzata, Università di Roma La Sapienza; M. Simmaco – Università di Roma La Sapienza
Negli ultimi anni abbiamo osservato un progressivo aumento dell’interesse sul tema della personalizzazione in medicina. Ad una crescente produzione scientifica, si è manifestata una maggiore attenzione anche della popolazione generale verso una nuova modalità di applicazione dei progressi ottenuti nello studio del genoma, con il conseguente rischio di una errata valutazione delle reali possibilità di questo nuovo approccio clinico. L’analisi di un numero di dati esponenzialmente crescente rende indispensabile l’integrazione strutturata, nel campo delle scienze omiche, di competenze bioinformatiche, statistiche ed epidemiologiche. Infatti, tutte le omiche sono rese possibili dall’evoluzione tecnologica e bio-informatica, per valutare, all’interno di un sistema complesso, un numero elevatissimo di dati (informazioni biologiche) da un singolo campione.
Solo la definizione di specifici percorsi basati sull’evidenza scientifica potrà ridurre il rischio di utilizzare strategie di medicina di precisione prive di evidenze sufficienti a garantire un reale e sostenibile vantaggio clinico rispetto alle modalità convenzionali attualmente in uso, rendendo, di fatto, insostenibile per qualsiasi modello di sistema sanitario affrontare i costi che ne deriverebbero, in assenza di sicuri miglioramenti assistenziali.
Prima di affrontare i possibili vantaggi per il Paziente e le difficoltà nell’implementazione di questi nuovi percorsi diagnostico-terapeutici è necessario fornire alcune definizioni, capaci di guidare anche i meno esperti in questi settori della Medicina.
Differenze tra medicina personalizzata e medicina di precisione
Molto spesso i termini di medicina personalizzata e medicina di precisione vengono considerati sinonimi di un approccio individualizzato alle diverse patologie.
Nel 2011, il National Research Council degli Stati Uniti ha definito la Medicina di precisione come l’adattamento di uno specifico trattamento alle caratteristiche individuali di un singolo paziente[1]. Ovviamente, ciò non significa che sia possibile creare un trattamento unico o identificare un singolo farmaco per ogni paziente, ma pone in evidenza quanto sia divenuto fondamentale tenere conto delle caratteristiche individuali di una singola persona per scegliere la terapia in grado di fornire i migliori risultati in termini di efficacia e di riduzione dell’incidenza di effetti collaterali. Spesso questo tipo di approccio viene definito come la Medicina delle 4 P (P4-medicine, predittiva, personalizzata, preventiva, partecipata), o anche la Medicina delle multiple P (Multiple-P Medicine), creando non poca confusione semantica, che non giova ad un corretto inquadramento delle sue possibilità e dei suoi limiti. Negli ultimi anni, la crescita delle conoscenze sulla biologia cellulare e la maggiore accuratezza delle tecniche di sequenziamento genomico, associata ad una esponenziale riduzione dei costi, ha permesso un rapido incremento dell’interesse e delle aspettative verso la medicina personalizzata[2], con due importanti conseguenze:- l’identificazione di specifici bersagli molecolari all’interno di cellule tumorali (alterazioni somatiche), che hanno portato allo sviluppo di agenti terapeutici mirati, progettati per interferire in modo specifico con eventi biologici chiave nei meccanismi di regolazione cellulare e nei rapporti tra diversi sistemi e apparati;
- la possibilità di misurare in modo ragionevolmente accurato, a livello del DNA di ciascun individuo (caratterizzazione germ-line), un numero sempre più elevato di indicatori biologici associati a specifici meccanismi cellulari, capaci di conferire una maggiore probabilità di contrarre specifiche patologie o una diversa capacità di risposta o tossicità ai trattamenti farmacologici.
Tuttavia, occorre ricordare che la differenza tra quanto è scritto nel genoma e l’effetto prodotto nei diversi soggetti risiede nelle differenze di espressione genica, nella qualità e nella quantità delle proteine tradotte dal genoma, e, dunque, nelle differenze nei metaboliti, ossia nei prodotti finali e intermedi delle reazioni chimiche che avvengono all’interno dell’organismo. Lo studio di questi sistemi a livello globale è ciò che chiamiamo scienze omiche, come la genomica (studio della sequenza, struttura e funzione del DNA), la trascrittomica (studio dei meccanismi di regolazione dell’espressione genica e dei suoi prodotti), la proteomica (studio delle proteine espresse, dei meccanismi che regolano la loro espressione, modificazione e funzione), la metabolomica (studio delle piccole molecole prodotte in seguito a reazioni biochimiche di molecole endogene ed esogene, come i farmaci), l’interattomica (studio delle connessioni e interazioni reciproche tra i pool di molecole biologiche), la microbiomica (studio dei microrganismi del microbiota), l’infettivomica (studio di microrganismi patogeni), la nutribiomica (studio per i cibi e/o i nutrienti), l’ambientomica (studio delle interzioni tra ambiente ie organismo, in generale), la palliomica (studio delle terapie di supporto individualizzate).
Le scienze omiche, per analizzare tipologie diverse di indicatori, devono necessariamente basarsi su un insieme di tecniche diverse che includono, fra le altre, immunoistochimica, immuno-feno-tipizzazione, ibridazione in situ, citogenetica, microarray, tecniche di biologia molecolare classiche, tecniche di sequenziamento (Sanger, next generation sequencing – NGS) e altre tecniche biochimiche tradizionali o innovative.
Sebbene il termine di Medicina di precisione abbia avuto una maggiore diffusione nella letteratura scientifica dopo la presentazione della National Precision Medicine Initiative da parte del Presidente Barack Obama nel suo State of the Union Speech del 2015, il termine di Medicina personalizzata mantiene una sua specifica valenza per indicare, al di là delle caratteristiche genomiche individuali o delle modificazioni indotte da uno specifico quadro patologico a livello cellulare, una metodologia capace di integrare in una valutazione olistica le caratteristiche fenotipiche (dalla etnia alle caratteristiche genealogiche, dagli indici antropometrici agli stili di vita) e genotipiche individuali, al fine di adattare qualitativamente, quantitativamente e in termini temporali un intervento sanitario di prevenzione, diagnosi o terapia alla necessità della singola persona.
La medicina personalizzata rappresenta un approccio complementare e non alternativo alla medicina tradizionale, basata su protocolli definiti per patologia, che rappresenta attualmente il percorso convenzionale caratterizzato dalla maggiore evidenza di prove scientifiche nei diversi percorsi sanitari in campo preventivo, diagnostico e terapeutico. Anche se le applicazioni cliniche di informazioni basate sulla personalizzazione degli interventi sanitari appaiono particolarmente innovativi ed interessanti, è necessario sottolineare come solo la definizione di specifici percorsi basati sulla evidenza scientifica potrà ridurre il rischio di utilizzare strategie di medicina personalizzata prima di disporre di evidenze sufficienti a garantire un reale e sostenibile vantaggio rispetto alle modalità standard attualmente in uso[3][4].In conclusione, le enormi possibilità rese disponibili dai progressi tecnologici consentono di avere a disposizione per ogni singolo paziente un numero di informazioni talmente elevato da rendere impossibile un loro impiego clinico, se non mediato da nuovi strumenti informatici di analisi e di aggregazione delle informazioni.
Il riconoscimento di alterazioni genomiche come guida alla identificazione di farmaci a bersaglio molecolare
Le attività diagnostiche su tessuti, cellule o derivati cellulari, soprattutto in campo oncologico, con utilizzo di indagini genomiche sono effettuate fin dagli anni 80 nelle strutture di Anatomia Patologica, di citogenetica e di biologia molecolare, presenti in tutto il territorio nazionale, utilizzando metodiche di immunoistochimica, di ibridizzazione in situ con metodica fluorescente (FISH) e cromogenica (CISH/SISH), alle quali negli anni si sono affiancate metodiche di sequenziamento Sanger o metodiche di spettrometria di massa e next generation sequencing (NGS).
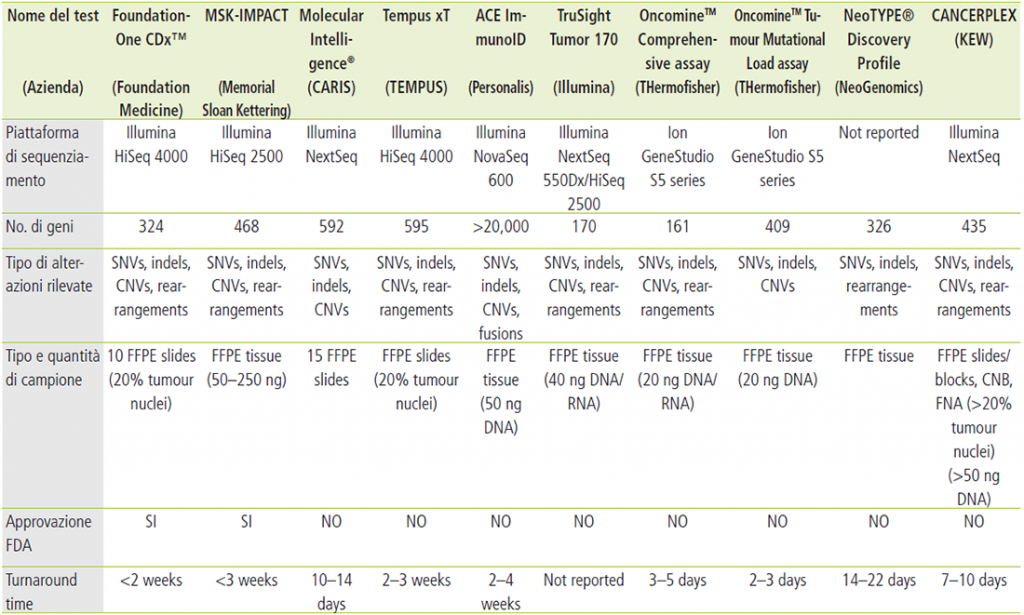
Tali attività rappresentano un’integrazione alla diagnostica anatomo-patologica e necessitano di specifiche competenze professionali e organizzative ormai ampiamente consolidate, grazie anche ad una proficua collaborazione tra diverse competenze professionali, nell’ambito dei Percorsi Diagnostici e Terapeutici Assistenziali (PDTA). La possibilità di interferire con queste alterazioni somatiche, presenti cioè all’interno delle cellule tumorali, ha consentito l’impiego di numerosi farmaci in specifiche patologie, con significativi vantaggi per i pazienti. Oltre la valutazione di singole alterazioni con uno specifico test, è possibile condurre indagini molecolari con pannelli di NGS. Queste tipologie di analisi sono estremamente complesse nella loro esecuzione e nella interpretazione dei risultati. Pertanto, queste indagini dovrebbero essere eseguite in laboratori di riferimento con un adeguato livello di dotazione tecnologica e con competenze specifiche nel settore[5]. La decisione di implementare test di NGS per la profilazione genetica ampia (comphehensive genetic profiling, CGP) nei laboratori diagnostici o di ricorrere a service esterni (a costi superiori per singolo esame) deve tenere in conto dei volumi di attività attesi, della esperienza del laboratorio e della eventuale partecipazione a programmi di ricerca. Esistono diverse tipologie di pannelli NGS per CGP che differiscono per tecnologie impiegate, dimensioni e tipologia di campioni utilizzati (tessuto o biopsia liquida), la cui scelta dipende dal quesito clinico a cui si vuole rispondere. Sono attualmente disponibili numerosi test commerciali per CGP, sia per analisi del tessuto che del cfDNA. La maggior parte dei test sono offerti mediante service, anche se recentemente alcuni pannelli per CGP sono stati sviluppati come kit per i laboratori di ricerca e diagnostici. I principali test disponibili sul mercato per analisi su tessuto e cfDNA sono indicati nelle successive tabelle (test su tessuto e test su cDNA), che tuttavia rappresentano un quadro parziale di un settore in continua evoluzione.Tabella 1. Alcune piattaforme per analisi di profilazione genomica su tessuto neoplastico
Tratto da Nicola Normanno – Antonio Marchetti – Giancarlo Pruneri: I test NGS. In Il nuovo modello mutazionale in oncologia, a cura di Nello Martini e Paolo Marchetti, Pensiero Scientifico Editore, 2019.
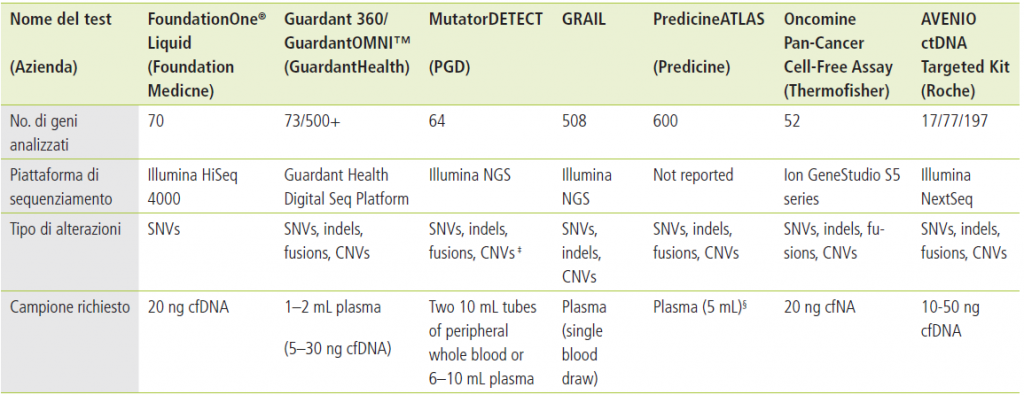
Tabella 2. Alcune piattaforme per analisi di profilazione genomica su DNA circolante
Tratto da Nicola Normanno – Antonio Marchetti – Giancarlo Pruneri: I test NGS. In Il nuovo modello mutazionale in oncologia, a cura di Nello Martini e Paolo Marchetti, Pensiero Scientifico Editore, 2019.
Il modello mutazionale
Per gli aspetti di ricerca clinica, per lo sviluppo di nuovi farmaci, per le procedure autorizzative a livello nazionale (AIFA) ed internazionale (EMA, FDA) e per gli aspetti organizzativi delle strutture assistenziali (oncologia ematologia – anatomia patologica – genetica molecolare), l’oncologia si è basata sul modello istologico, secondo una sequenza che prevede:
- la localizzazione del tumore
- la caratterizzazione istologica
- la valutazione dell’eventuale presenza di mutazioni geniche
- l’identificazione di farmaci a bersaglio molecolare
- la applicazione delle condizioni di prescrizione e di rimborso
Lo scorso anno, L’FDA ha autorizzato l’impiego di un farmaco, il pembrolizumab, per il trattamento di tumori solidi metastatici non resecabili con un’elevata instabilità dei microsatelliti (MSI – H) o un deficit del mismach repair (dMMR), secondo una procedura definita di agnostic approval, in quanto l’autorizzazione all’impiego del farmaco si basa sul riconoscimento di una specifica mutazione, indipendentemente dalla sede del tumore. A questa prima approvazione è seguita quella del larotrectinib per neoplasie maligne in pazienti con il riarrangiamento del gene NTRK e dell’entrectinib per il trattamento di tumori solidi in pazienti con riarrangiamento dei geni NTRK 1,2 3 – ROS1.
In conclusione, a livello regolatorio, sta confermandosi la possibilità di una autorizzazione all’impiego clinico di un farmaco in base ad un modello non istologico ma mutazionale, sia pure in presenza di dati preliminari, non consolidati e che sono sottoposti ad ulteriore conferma, fondato sulla mutazione somatica e su un modello diverso da quello istologico che fino ad ora ha governato la ricerca clinica, le decisioni regolatorie e la partica oncologica.
La farmacogenomica come aspetto fondamentale della riconciliazione terapeutica
La ricerca farmaceutica ha permesso, nel corso degli anni, di sviluppare una gran quantità di farmaci per la cura di moltissime patologie. Ogni farmaco tipicamente è rivolto alla cura (eradicazione) o al rallentamento della evoluzione di una specifica malattia o alla riduzione dei sintomi collegati, pur in presenza di un numero diverso di effetti collaterali, variabili per numero ed intensità in ogni singolo soggetto. Tuttavia, oltre agli effetti collaterali di un singolo farmaco, ampiamente descritti nelle schede tecniche e nei foglietti illustrativi dei singoli prodotti, i pazienti che assumono più farmaci possono subire effetti negativi a seguito delle loro interazioni o della diversa capacità dell’organismo di metabolizzare (cioè, eliminare) i farmaci, con conseguenti modificazioni della loro efficacia e/o della comparsa di ulteriori effetti collaterali anche gravi. Sebbene sia obbligatorio riportare nella scheda tecnica di un singolo farmaco le sue interazioni note con altri principi attivi o con il cibo, è molto complesso e di difficile applicazione pratica analizzare le interazioni tra molti farmaci. Infatti, il rischio di una interazione farmacologica negativa aumenta con il numero di patologie presenti e, conseguentemente, con il numero di farmaci necessario a trattare ciascuna patologia. Per esempio, alcuni farmaci anti-infiammatori, usati a scopo antalgico, possono avere influenze negative sulla funzionalità renale o sulla pressione sanguigna e debbono essere utilizzati con molta cautela in pazienti che già presentano ipertensione arteriosa o malattie renali. Infine, mentre sono noti e, in genere, attentamente valutate le interazioni tra due farmaci, molto più complesse, e scarsamente valutabili in clinica, sono le interazioni tra più farmaci.
Alla luce delle più recenti acquisizioni in campo analitico e clinico, appare importante approfondire le implicazioni di queste osservazioni, sottolineando la necessità di un approccio multidisciplinare necessario al corretto utilizzo di dati farmacogenomici. Sebbene possa essere difficile individuare il rapporto costo-beneficio dell’analisi di singoli geni/polimorfismi con effetto sulla risposta a determinati farmaci, proprio una “visione sistemica” può facilitare l’individuazione degli ambiti clinici in cui l’approccio farmacogenomico risulta ormai praticamente imprescindibile.
Consideriamo, infatti, la traslazione verificatasi tra “Genetica medica” e “Genomica”, che riflette quella ormai esistente tra “test genetico” e “test genomico”. La Genomica rappresenta l’evoluzione della Genetica medica classica, prevalentemente incentrata sullo studio di uno o pochi geni francamente associati a malattie ereditarie. In Genomica moltissime variazioni comuni (polimorfismi) nella sequenza di DNA, ovvero presenti con buona frequenza (>1%) nella popolazione sana sono imprescindibili per comprendere la fisiopatologia e l’eziologia di una molteplicità di malattie propriamente non a carattere “genetico”. Nella genomica si ha una vera e propria “visione di sistema”, per cui il medico ha cominciato a concepire la complessità dei fattori endogeni ed esogeni che influenzano la comparsa e il decorso non solo di malattie, (genetiche e non) ma anche la risposta ai trattamenti medici, in termini di efficacia e tossicità.
Simile la differenza tra test (farmaco)genetici (uno o poche varianti analizzate sia su DNA germinale che somatico) e test (farmaco)genomici (da decine a centinaia di varianti analizzate): quest’ultimo tipo di test ha probabilmente un costo complessivo maggiore, ma non se si calcola il costo per singolo analita (variante).
Molto spesso i pazienti oncologici sono affetti anche da altre patologie croniche e vengono seguiti da specialisti diversi. Quando viene meno una stretta interazione professionale nell’ambito di gruppi interdisciplinari o quando il paziente assume farmaci in auto-prescrizione, è possibile che si verifichino interazioni farmacologiche anche gravissime o perdita di efficacia delle terapie oncologiche. Pertanto, la riconciliazione terapeutica, intesa come la prescrizione di farmaci sotto un’unica regia, che tenga conto delle diverse prescrizioni specialistiche, è di fondamentale importanza.
Oltre 200 farmaci contengono nella loro scheda tecnica specifiche indicazioni di farmacogenomica[6] e circa un terzo di queste riguardano farmaci oncologici. La farmacogenomica può rivestire un ruolo importante nella identificazione di soggetti con diverso grado di risposta ai trattamenti farmacologici, riducendo l’incidenza di effetti collaterali anche gravissimi ed ottimizzando il dosaggio dei farmaci (non solo oncologici).Per paziente politrattato si intende un paziente che assume in modo continuativo più farmaci contemporaneamente a seguito della presenza di diverse malattie, ciascuna delle quali richiede uno specifico trattamento farmacologico. L’Organizzazione Mondiale della Sanità (WHO) considera politrattati i pazienti che assumono da 5 a più farmaci diversi al giorno.
Nonostante gli Enti regolatori (FDA, EMA, AIFA, per citarne solo alcuni) abbiano indicato specifici percorsi clinici per valutare le interazioni negative tra farmaci in pazienti politrattati per la contemporanea presenza di malattie diverse, non esistono ancora modalità di semplice utilizzo in clinica per una valutazione olistica di questi pazienti[7], con conseguenze significative in termini di spese e sofferenze evitabili per il Sistema sanitario Nazionale e per i pazienti. I pazienti anziani sono ovviamente quelli maggiormente esposti a questi rischi, non solo perché maggiore è la probabilità di essere afflitti da patologie croniche (ad esempio, diabete, obesità, ipertensione, patologie osteoarticolari) che richiedono ciascuna un diverso trattamento farmacologico (rendendoli politrattati secondo la WHO), ma anche perché con l’avanzare dell’età si modificano le capacità metaboliche dell’organismo con conseguenti modificazioni della farmacocinetica e della farmacodinamica dei diversi composti assunti a cui seguono ulteriori effetti collaterali. Da uno studio effettuato di Morgan, T.K., et al.[8] è emerso che circa il 45% delle persone al di sopra di 50 anni assume regolarmente più di 5 tipi di farmaci diversi. In italia, Onder et al.[9] hanno documentato come su oltre 12 milioni di pazienti ultra-sessantacinquenni ve ne siano oltre 7 milioni che assumono da 5 a oltre 10 farmaci al giorno.L’assunzione di svariati farmaci, tra loro di eterogenea natura, può portare a delle interazioni. Al crescere del numero di farmaci assunti da una persona, la gestione dell’interazione dei farmaci diviene sempre più critica, perché con il crescere del numero di farmaci assunti contemporaneamente il rischio di interazioni negative anche rilevanti aumenta significativamente. A volte, la presenza di un effetto collaterale di un farmaco non viene riconosciuto o richiede uno specifico trattamento, mediante la somministrazione di un ulteriore farmaco. In questo contesto assistenziale, spesso risulta difficile comprendere se un determinato sintomo deriva dal mancato controllo di una delle patologie da cui è affetto il paziente o da una interazione negativa tra i farmaci, per incompatibilità note o per modificazioni nel loro metabolismo dovute proprio ad alterazioni di un singolo soggetto. Interazioni negative che si possono sviluppare tra farmaci assunti contemporaneamente possono allungare i tempi di cura di una o più patologie, influenzare gli effetti di altri farmaci al punto tale da renderli inutili per la cura della patologia per la quale originariamente erano previsti e possono portare a ospedalizzazioni del paziente o ancora all’insorgenza di nuove patologie. Tutto questo comporta una riduzione dello stato di salute complessivo dell’individuo e in molti casi un inutile dispendio economico.
Sono noti alcuni database, tra i quali “Transformer”, reperibile al sito http://bioinformatics.charite.de/transformer, che contiene dati di circa 2800 farmaci, con circa 60.000 indicazioni di possibili conflitti tra farmaci, di cui 5.500 interazioni relative al metabolismo di fase I (enzimi citocromo P450 o CYP, che rappresenta il principale meccanismo di detossificazione dell’organismo da farmaci e che è uno dei fattori che più concorrono a definire la variabilità del rapporto dose/risposta in soggetti differenti che assumono un medesimo farmaco) e/o di fase II (altri enzimi), e che comprende inoltre dati relativi ai trasportatori farmacocinetici, interazioni alimentari (circa 350) e oltre 100.000 riferimenti bibliografici.
Come già accennato, infatti, diversi fattori tra cui l’etnia del soggetto e la tipologia del farmaco influiscono sul verificarsi o meno di interazioni sulla combinazione di farmaci diversi che il soggetto assume. Ad esempio, farmaci quali benzodiazepine o altri farmaci psicoattivi possono produrre interazioni avverse in relazione alla dose alla quale sono assunti.
Sono disponibili sul WEB alcuni programmi volti a valutare le interazioni farmacologiche (Drug Interaction Checker: www.webmd.com/interaction-checker/default.htm. www.rxlist.com/drug-interaction-checker.htm. https://reference.medscape.com/drug-interactionchecker. www.drugs.com/drug_interactions.html). Tuttavia, questi programmi si limitano ad identificare possibili interazioni di un farmaco con un altro presente nella lista inserita dal medico, fornendo un lungo elenco di possibili interazioni negative. Dopo aver letto tutta la sequenza di interazioni, il medico deve provvedere a riformulare la prescrizione, sottoponendola di nuovo al sistema in una iterazione che può richiedere anche moltissimo tempo, incompatibile con le esigenze cliniche. Infine, in questi programmi non vengono mai valutate le caratteristiche cliniche di un singolo soggetto o le sue patologie concomitanti capaci di modificare la biodisponibilità del farmaco.
Altri sistemi di gestione delle interazioni tra farmaci permettono, invece, un’ottimale analisi delle interazioni e consentono di determinare in modo efficace e rapido la migliore combinazione di farmaci da assegnare ad un soggetto affetto da più patologie contemporaneamente, per le quali siano necessari trattamenti protratti nel tempo.
Sebbene siano disponibili numerosissimi lavori scientifici che caratterizzano la diversa attività e tossicità di trattamenti farmacologici in funzione di specifici polimorfismi nei geni coinvolti nel metabolismo dei farmaci, è necessario acquisire ulteriori informazioni, ad esempio in tema di farmaco-economia, per definire i vantaggi reali di un approccio innovativo nei pazienti politrattati. Un test farmacogenomico ideale deve contenere tutte le varianti geniche ben note per il loro coinvolgimento nella farmacocinetica e nella farmacodinamica (enzimi metabolici, trasportatori, bersagli molecolari). L’informazione derivata deve coerentemente essere integrata con una analisi delle interazioni farmaco-farmaco, poiché le variazioni genomiche sono notoriamente in grado di modificare l’interazione farmaco-farmaco attesa.
In conclusione, è indispensabile caratterizzare gli ambiti dove è vantaggioso utilizzare l’approccio farmacogenomico, avendo cura di individuare tutte le fonti di spesa sanitaria derivate dalla mancata efficacia delle terapie o dallo sviluppo di tossicità, eventi che possono essere efficientemente ridotti con la farmacogenomica.
La network medicine
La visione contemporanea della malattia risale al XIX secolo e si basa in gran parte sulla correlazione clinicopatologica osleriana, ossia l’associazione tra dato clinico e quello istologico. Tale semplificazione è stata utile per i clinici del passato in quanto stabilisce modelli di malattia che limitano il numero dei potenziali fenotipi patologici (pato-fenotipi). Tuttavia, classificare la malattia in questo modo risulta inadeguato e riduzionistico nell’era post-genomica. Infatti, anche la più semplice malattia geneticamente determinata è complessa nella sua espressione e può presentare pato-fenotipi diversi. Un semplice esempio è rappresentato dall’anemia falciforme, malattia monogenica mendeliana causata da una mutazione puntiforme della catena beta dell’emoglobina, che ne modifica l’affinità per l’ossigeno, promuovendone la polimerizzazione in condizioni ipossiche. Nonostante la predizione mendeliana, questo semplice fenotipo biochimico si associa a multipli pato-fenotipi: individui con anemia falciforme possono presentare crisi dolorose, osteonecrosi, dolore toracico acuto, ictus, anemia severa o anemia lieve. Le ragioni alla base di questi diversi pato-fenotipi clinici spaziano dalla presenza di geni modificanti la malattia (ad esempio, l’emoglobina F) a influenze ambientali (ad es., l’ipossia). Osservando che anche la più semplice malattia geneticamente determinata è complessa nella sua espressione, si deve considerare l’importanza dei contesti genomici (deterministici) e ambientali (stocastici) entro i quali la malattia evolve. Ne risulta che la malattia è raramente la semplice conseguenza di un’anomalia in un singolo prodotto genico, ma, piuttosto, è un riflesso dei processi pato-biologici (deterministici e stocastici) che interagiscono in una rete complessa per produrre un pato-fenotipo.
Entro tale contesto nasce la network medicine, come approccio di sistema che sfida il paradigma della malattia esistente e cerca di identificarne meccanismi pato-biologici, vedendoli da un punto di vista olistico[10] [11] [12]. La patobiologia dei sistemi è definita come la scienza dell’integrazione di dati genetici, genomici, biochimici, cellulari, fisiologici e clinici con il fine di creare una rete che può essere utilizzata per definire predittivamente le manifestazioni della malattia e la risposta alle terapie.Per meglio comprendere la medicina di rete, è importante revisionare alcuni semplici concetti che riguardano la teoria delle reti.
Le reti sono costituite da due elementi essenziali: i nodi e i legami tra i nodi. Qualsiasi rete può essere vista come un insieme di nodi interconnessi, la cui distribuzione può variare da casuale ad altamente clusterizzata. Le reti biologiche non sono reti casuali, ma i nodi costituenti (geni, RNA regolatori, proteine, metaboliti) si organizzano in cluster. I nodi altamente connessi di tali reti sono definiti hubs. I subnetwork o moduli che possono identificarsi all’interno della rete sono responsabili di specifici processi pato-biologici o di una specifica malattia. Un modulo di malattia è definito come un gruppo di componenti di rete che contribuisce a un fenotipo, la cui rottura porta a un particolare pato-fenotipo. Più precisamente, un modulo di malattia rappresenta una sottorete nella rete molecolare complessiva che riflette un insieme unico di interazioni, sia prossimali che remote, che contribuiscono a un fenotipo anomalo, quando uno o più dei suoi componenti risulta essere disfunzionale.
Nell’ambito di una malattia, considerando la sua rappresentazione di rete, un gene, una proteina o un metabolita specifici possono partecipare a diversi moduli. Nonostante la gran mole di dati accumulati negli ultimi decenni, il numero delle interazioni funzionalmente rilevanti tra i componenti della rete resta in gran parte sconosciuto. Tale interconnessione a livello subcellulare implica che l’impatto di una alterazione genetica non si ripercuota solo sul prodotto di tale gene, ma che l’effetto diffonda lungo i legami della rete, alterando l’attività dei prodotti genici che altrimenti non sarebbero interessati. Pertanto, l’impatto fenotipico di un difetto genico/proteico dipende dal contesto di rete. Inoltre, per comprendere la migliore espressione della malattia, è necessario non solo definire l’architettura o la topologia della rete della malattia nel contesto dell’universo delle reti molecolari in una cellula o organismo, ma anche per esplorarne le risposte dinamiche alle perturbazioni. Poiché nel corso degli scorsi decenni abbiamo raccolto molti dati sulle interazioni molecolari umane, i network al momento più conosciuti e studiati sono quelli molecolari, ossia network di interazione proteica (i cui nodi sono proteine legate tra loro da interazioni fisiche), network metabolici (i cui nodi sono costituiti da metaboliti, legati tra loro se coinvolti nella stessa reazione biochimica), network regolatori (i cui legami sono costituiti, a livello trascrizionale, dalla interazione regolatoria tra fattore trascrizionale e gene, mentre a livello post trascrizionale, i legami si stabiliscono tra chinasi e loro substrati) e network di RNA (i cui nodi sono costituiti da microRNA e siRNA). I moduli di malattia, all’interno di tali reti, possono essere identificati usando metodi bioinformatici o sperimentali, nei cui dettagli non ci addentreremo per la complessità della tematica.
Nell’era della medicina di precisione, il target molecolare “disfunzionale” è oggetto di grande interesse sia per la classificazione molecolare delle malattie che per l’approccio terapeutico. In particolare, nell’ambito dell’oncologia, dall’identificazione di c-kit nel secolo scorso, fino ai giorni nostri, gli investimenti finanziari da parte delle aziende farmaceutiche sono stati rivolti all’introduzione sul mercato di farmaci selettivi in grado di inibire il prodotto genico difettoso. Tuttavia, gli eventi mutazionali non sono tutti ugualmente rilevanti ed il loro impatto clinico è funzione anche del contesto che li ospita.
Alcune mutazioni sono driver importanti nel processo di cancerogenesi e, pertanto, sono in grado di guidare l’evoluzione clonale della malattia. Altre mutazioni, definite passengers, sono eventi mutazionali clinicamente e fenotipicamente neutrali nello sviluppo e progressione tumorale. Distinguere tali mutazioni costituisce un primo elemento nell’identificazione della terapia, ma non è sufficiente. Infatti, il livello genomico è solo un primo piano nell’edificio della conoscenza oncologica e necessita l’integrazione di ulteriori livelli (tra cui trascrittomica, epigenomica, proteomica, metabolomica). Inoltre, il contesto, ossia il tessuto nel quale tali alterazioni si manifestano, e la sequenzialità degli eventi (ad oggi terreno quasi inesplorato), potrebbero essere elementi rilevanti nella scelta terapeutica.
Esemplifichiamo il concetto di contesto con un esempio: BRAF è un gene driver, codificante per una chinasi coinvolta nella via di ERK, via fondamentale per la crescita, differenziazione e sopravvivenza cellulare. Le mutazioni di BRAF sono presenti in circa l’8% dei tumori umani, con una differente frequenza per istotipo (contesto). In particolare, le mutazioni V600 di BRAF si verificano in circa il 100% delle leucemie a cellule capellute, nel 50% dei melanomi cutanei, nel 37% dei pazienti con carcinoma papillifero della tiroide, nel 5-8% dei pazienti con adenocarcinoma del colon-retto metastatico, nell’1-2% degli adenocarcinomi polmonari. Spesso, la presenza di tale mutazione è associata ad una prognosi sfavorevole: malattia più aggressiva, minore tasso di risposte alla terapia, più breve sopravvivenza globale. Il vemurafenib, inibitore della chinasi BRAF, ha mostrato eccellenti risposte quando utilizzato in alcuni tumori (100% di risposta nella leucemia a cellule capellute), buone risposte nel melanoma, attività intermedia nell’adenocarcinoma polmonare e nel tumore della tiroide, risposta modesta nel tumore del colon-retto. Tali dati suggerirebbero la necessità di non trascurare l’humus (contesto) nel quale il tumore si instaura.
La sperimentazione clinica attraverso i basket trials, avvalendosi del solo dato molecolare, agnostico dell’istologia, costituisce un tentativo di adeguarsi ad uno scenario in rapida trasformazione, carico di dati genomici. Sebbene dei traguardi in termini di risposte a breve termine siano stati raggiunti, proseguire esclusivamente lungo questa strada, non tenendone conto dei limiti, rappresenterebbe una ricaduta nell’approccio riduzionistico del secolo scorso, con la mera sostituzione dell’espressione associazione clinico-patologica con associazione clinico-molecolare. Servirsi di tale strategia è giustificato al momento solo dall’incapacità di integrare, a livello clinico e sperimentale, i dati omici raccolti e dalla mancanza di serie temporali di tali dati.
Identificare la medicina di precisione con il target molecolare da spegnere nel tumore, ignorando ogni altro elemento, è un errore da evitare e superare, per quanto consapevoli della carenza di validi strumenti alternativi nella sperimentazione farmacologica contro il cancro.
Avvalersi della network medicine, attraverso la sua visione di sistema, permetterà di avvicinarsi alla complessità della rete cancro, unendo differenti serie di dati. Inoltre, la realizzazione di un network temporale, composto da una serie di network statici rappresentativi di un dato momento, potrà aiutare a comprendere l’evoluzione delle alterazioni e interazioni molecolari che si sviluppano durante un dato processo, superando anche il limite dei legami permanenti. L’effetto dei social network sul comportamento e le sue conseguenze sull’espressione della malattia aggiunge un’ulteriore dimensione agli approcci sistemici alla malattia umana. Infine, la complessità del microbioma, le sue interazioni con il genoma dell’ospite umano e le conseguenze patobiologiche della sua perturbazione sono un’altra area di indagine. Il lavoro è lungo ed impegnativo in quanto prevede la necessità di creare task force di collaborazione tra professionisti con competenze diverse (cliniche, molecolari, bio-mediche, informatiche).
La complessità del modello mutazionale e la creazione del Molecular Tumor Board (MTB)
Il modello mutazionale che si affianca al modello istologico tradizionale risulta molto complesso e impatta su molte variabili scientifiche, organizzative, etiche, di privacy e di riorganizzazione di servizi.
Uno dei punti chiave di una nuova governance correlata al modello mutazionale in oncologia ed ematologia si basa sulla istituzione di Molecular Tumor Board – MTB, per gestire la complessità e la comprensione dei test di genomic profiling, la scelta dei farmaci (o delle combinazioni) attivi che possono essere già disponibili e rimborsati, oppure disponibili, ma non registrati per la specifica indicazione (off-label) o in fase di sperimentazione clinica.
Un network di MTB, possibilmente accreditato da AIFA, secondo criteri e procedure trasparenti per la composizione, le attività, la tracciabilità e la elaborazione dei dati può rappresentare il nuovo strumento di gestione del modello mutazionale nella normale pratica clinica.
L’estrema complessità della gestione del modello mutazionale in oncologia richiede in maniera imprescindibile la attivazione di gruppi interdisciplinari, in cui siano integrate le competenze di oncologia, ematologia, biologia molecolare, genetica, anatomia patologica, farmacia, bioinformatica e bioetica per poter governare i processi clinici e decisionali di appropriatezza, ovvero:
- la stadiazione e le linee di trattamento del tumore secondo Linee Guida nazionali e internazionali
- le mutazioni genetiche e le mutazioni driver che codificano per lo sviluppo del tumore, il pathway delle mutazioni e il valore predittivo del tumor molecular burden
- l’analisi e la valutazione del report del genomic profile – NGS del paziente
- la scelta dei farmaci oncologici più appropriati in commercio o in fase di registrazione o per indicazioni attualmente off label
- la raccolta sistematica dei dati clinici e della genomica per consolidare le conoscenze e per sviluppare attività di ricerca in un settore in cui le evidenze non sono ancora consolidate, pur esprimendo un valore aggiuntivo clinicamente rilevante.
L’integrazione dei modelli: un diritto per tutti i Pazienti, in una prospettiva di equità, universalità e sostenibilità.
La grande disponibilità di dati sul genoma ha aperto nuove opportunità che rendono nel futuro prossimo più facili e fruttuosi i processi propri della medicina di precisione. Infatti, tramite l’individuazione di varianti geniche comuni implicate nelle patologie e nella risposta ai farmaci è possibile migliorare e ampliare la comprensione delle vie biochimiche coinvolte, aprendo nuove possibilità di diagnosi e cura.
La possibilità di effettuare in maniera sempre più estesa test di profilazione genica, a costi progressivamente decrescenti, pone al Sistema Sanitario Nazionale l’imprescindibile esigenza di definire con chiarezza regole di accesso ai test, prima che di definizione del farmaco da prescrivere per una determinata alterazione molecolare.
Il Ministero della Salute, attraverso un tavolo per l’Attuazione dell’Intesa Stato-Regioni del 26.10.2017 concernente il Piano per l’innovazione del sistema sanitario basata sulle scienze omiche, sta definendo concretamente i percorsi di questa nuova rivoluzione.
In questo contesto normativo ed organizzativo è necessario valutare l’opportunità di istituire a livello regionale dei tavoli di discussione (Molecular Tumor Board), che siano in grado di definire, in maniera condivisa e omogenea su tutto il territorio nazionale, le diverse modalità di accesso a queste enormi potenzialità che la ricerca ha posto al servizio dei cittadini malati.
Note
1. ^ National Research Council of the National Academies, Towards precision medicine, The National Academies Press, 2011. 2. ^ Prasad V, Fojo T, Brada M. Precision oncology: origins, optimism, and potential. Lancet Oncol. 2016;17(2). 3. ^ Salgado R, Moore H, Martens JWM, et al; IBCD-Faculty. Societal challenges of precision medicine: Bringing order to chaos. Eur J Cancer. 2017;84:325-334. 4. ^ Hayes DF. Precision Medicine and Testing for Tumor Biomarkers-Are All Tests Born Equal? JAMA Oncol. 2018;4(6):773-774. 5. ^ N Normanno, A Marchetti, G Pruneri I test NGS. In Il nuovo modello mutazionale in oncologia, Pensiero Scientifico Ed., 2019. 6. ^ https://www.fda.gov/Drugs/ScienceResearch/ucm572698.htm 7. ^ Loscalzo J, Barabasi A-L. Systems biology and the future of medicine. WIREs Syst Biol Med 2011; 3: 619-27. 8. ^ A national census of medicines use: a 24-hour snapshot of Australians aged 50 years and older.” Med J Aust, 2012, 196, 50-53. 9. ^ Prescription Drug Use Among Older Adults in Italy: A Country-Wide Perspective, JAMDA 2014: 15,7: 531-535. 10. ^ J. Loscalzo, AL. Barabasi. Systems Biology and the Future of Medicine. Wiley Interdiscip Rev Syst Biol Med. 2011 Nov; 3(6): 619–627. 11. ^ AL. Barabási, N. Gulbahce, J. Loscalzo. Network Medicine: A Network-based Approach to Human Disease. Nat Rev Genet. 2011 Jan; 12(1): 56–68. 12. ^ J. Menche et al. Uncovering disease-disease relationships through the incomplete human interactome. Science. 2015 Feb 20; 347(6224): 1257601.Scarica questo capitolo in PDF:
11° Rapporto - Capitolo 9
La Medicina Personalizzata oltre i bersagli molecolari: dalla parte dei Pazienti